Introducción a sabinaNSDM: Un nuevo paquete de R para mejorar los modelos de la distribución de especies basado en modelos jerárquicos anidados espacialmente
Teresa Goicolea, Alejandra Zarzo
Los Modelos de Distribución de Especies (SDMs, por sus siglas en inglés) son herramientas esenciales para que científicos y especialistas de la conservación puedan predecir dónde es probable encontrar especies, dónde han existido en el pasado y dónde podrían aparecer en el futuro. Ante problemas urgentes como el cambio climático y la pérdida de biodiversidad, generar predicciones precisas es más importante que nunca para identificar áreas clave para aplicar medidas de conservación. Sin embargo, los SDMs a menudo tienen problemas de precisión, especialmente debido al truncamiento de nicho y problemas de extrapolación ambiental.
Ahí es donde se encuadra el nuevo paquete de R sabinaNSDM. Diseñado por nuestro equipo de investigación SABINA, este paquete utiliza un nuevo enfoque para construir SDMs, conocido como modelos jerárquicos anidados espacialmente (N-SDMs). Al combinar patrones globales a gran escala con características regionales más finas, sabinaNSDM permite generar predicciones más precisas de las distribuciones de las especies. Esto convierte al nuevo paquete en un recurso potente para la planificación de la conservación y la investigación ecológica.
El problema con los SDMs tradicionales
Los SDMs estándar presentan un conjunto de limitaciones. La mayoría de los modelos se clasifican en una de dos categorías: regional o global.
- Los modelos regionales se centran en áreas específicas, como un país o una región. Si bien pueden ofrecer información detallada sobre las condiciones locales, carecen de la perspectiva ambiental más amplia que da forma a la distribución de una especie. Esto lleva a lo que se llama el truncamiento de nicho, donde los modelos no consideran el rango completo de condiciones que una especie experimenta a lo largo de su distribución (es decir, el nicho ecológico). Estos modelos restringidos espacialmente también sufren de una mayor proporción de condiciones no análogas, lo que genera problemas al proyectar a otras áreas (por ejemplo, para predecir la expansión de especies invasoras) o períodos (para predecir el impacto del cambio climático en la distribución de especies).
- Por otro lado, los modelos globales cubren todo el rango de una especie, pero a menudo se basan en datos poco detallados y de baja resolución. Además, suelen basarse únicamente en variables bioclimáticas, ya que otros factores ambientales no están disponibles a gran escala, y de datos de especies imprecisos. Como resultado, carecen de los detalles finos necesarios para predicciones localizadas precisas.
La solución: Modelos de Distribución de Especies Anidados (N-SDMs)
Los SDMs jerárquicos anidados espacialmente (N-SDM) abordan estos problemas combinando la perspectiva amplia de los modelos globales con el detalle fino de los modelos regionales para obtener lo mejor de ambos. Los modelos globales proporcionan una visión general, capturando el nicho ecológico completo de una especie a lo largo de su rango y teniendo en cuenta factores como el clima a una resolución gruesa. Luego, los modelos regionales se centran en detalles más finos, como el uso del suelo o las condiciones de microhábitat y datos de distribución de especies más precisos, que suelen estar disponibles para áreas más pequeñas, como a nivel nacional. Estos detalles finos son críticos para hacer predicciones precisas y de alta resolución.
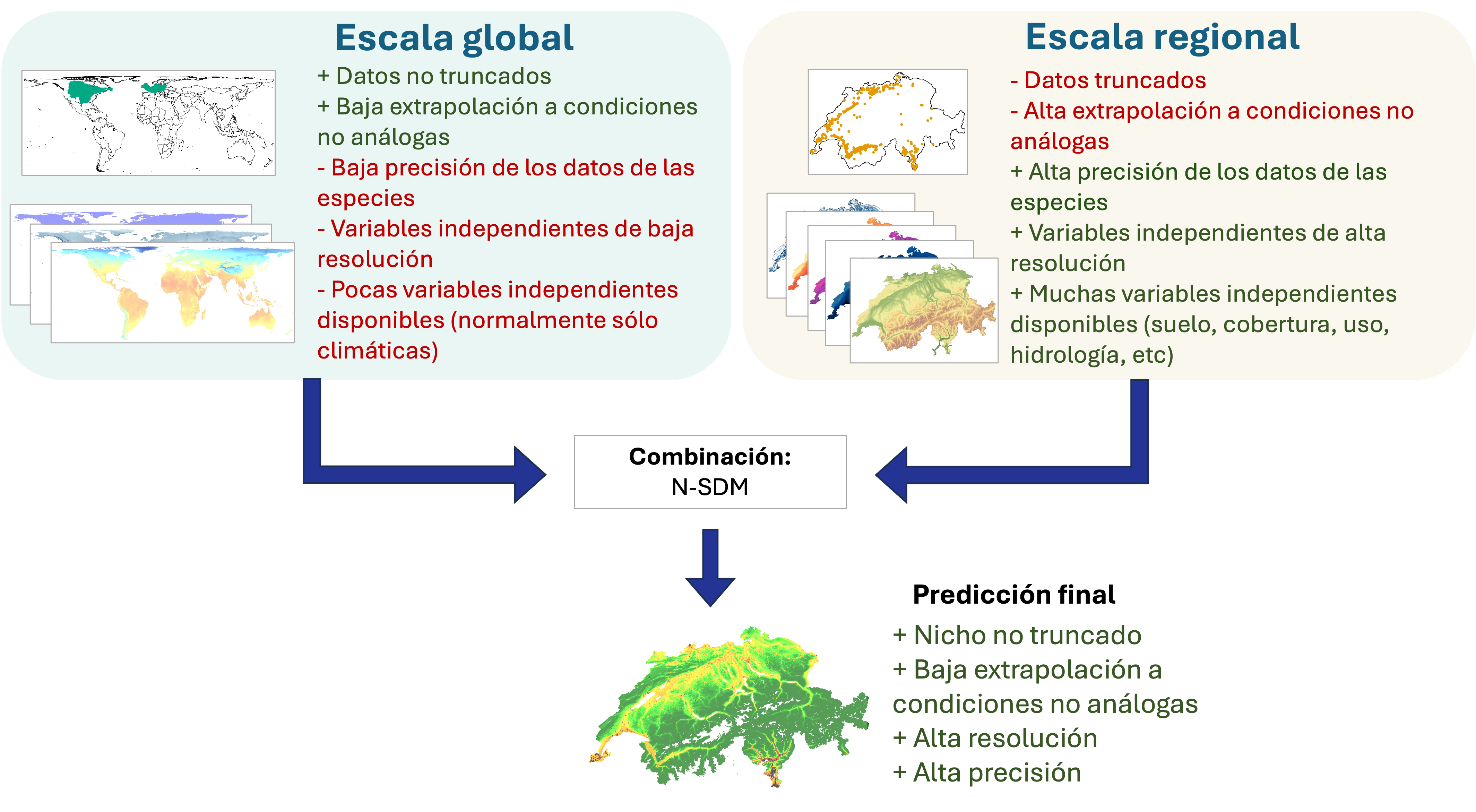
Figura. Ventajas (en verde) y limitaciones (en rojo) de los modelos tradicionales de distribución de especies (tanto a escala global como regional), en comparación con los beneficios de combinarlos en un Modelo de Distribución de Especies Jerárquico Anidado Espacialmente (N-SDM).
Características clave del paquete sabinaNSDM
sabinaNSDM está diseñado para hacer que este enfoque N-SDM sea más accesible para investigadores y especialistas de la conservación. Aquí están algunas de sus características clave:
- Generar N-SDMs: El paquete combina modelos globales y regionales.
- Diferentes estrategias de anidamiento: Los usuarios pueden elegir entre dos métodos para combinar modelos: el enfoque de covariables, que utiliza la salida del modelo global como entrada para el modelo regional, o el enfoque múltiple, que promedia las predicciones global y regional.
- Modelos de consenso: sabinaNSDM utiliza modelos de consenso, una técnica que combina múltiples algoritmos estadísticos para aumentar la fiabilidad y precisión de las predicciones.
- Flujo de trabajo integral: el paquete es una herramienta que integra (a) la generación de datos de fondo; (b) la preparación y el filtrado espacial de ocurrencias de especies (y ausencias si están disponibles); (c) selección de covariables ambientales; y (d) calibración, evaluación y proyección de N-SDMs.
- Eficacia demostrada: En un estudio aplicado sobre 77 especies de árboles y arbustos en la Península Ibérica, sabinaNSDM superó a los SDMs tradicionales, ofreciendo predicciones más precisas de las distribuciones de estas especies.
- Código abierto y fácil de usar: sabinaNSDM está disponible de manera gratuita en GitHub, y estamos trabajando para que esté disponible en CRAN. Este paquete está diseñado para ser fácil de usar, lo que lo hace accesible para ecólogos y especialistas de la conservación con diversos niveles de experiencia en programación.
Impacto en el mundo real
La capacidad de modelar con precisión las distribuciones de especies tiene consecuencias en el mundo real, y las capacidades mejoradas de modelado de sabinaNSDM pueden desempeñar un papel crucial para orientar de manera más efectiva los esfuerzos de conservación. Por ejemplo, el paquete puede predecir cómo el cambio climático podría alterar la distribución de las especies, guiar programas de restauración para señalar áreas con el mayor potencial para proteger la biodiversidad, o anticipar la propagación de especies invasoras. Una de nuestras aplicaciones clave ha sido crear un geoportal que muestra la distribución prevista de 200 especies de plantas leñosas en España bajo condiciones actuales y bajo cuatro escenarios climáticos futuros. El geoportal ofrece diversas aplicaciones prácticas como la generación de listas de los arbustos y árboles con la mayor idoneidad para ubicaciones específicas. Esto puede ayudar a informar los esfuerzos de restauración al identificar las especies más propensas a prosperar tanto ahora como en el futuro. sabinaNSDM ya ha demostrado su potencial en nuestro trabajo, y nos entusiasma ver cómo otros investigadores y especialistas de la conservación lo utilizan en sus proyectos.
Empieza a usar sabinaNSDM
Si estás interesado en probar sabinaNSDM, puedes descargar el paquete y explorar sus características en nuestro repositorio de GitHub. Para una inmersión más profunda en su funcionamiento, consulta nuestro artículo publicado en Methods in Ecology and Evolution. También hemos incluido material suplementario y tutoriales para ayudarte a empezar a trabajar con modelos de una o varias especies. Si estás interesado en saber más sobre sabinaNSDM o tienes alguna pregunta, no dudes en ponerte en contacto.



